EvoGrid
Un sistema informático pretende simular las condiciones de los océanos primitivos en miles de ordenadores y ver si aparecen formas autoorganizadas de vida digital.
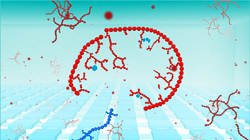
|
|
|
La diferencia entre países que apoyan la ciencia y los países que no lo hacen no depende tanto de la calidad de la clase política, sino de la calidad de la sociedad sobre la que ésta se asienta.
Que la sociedad de un país tenga inquietudes científicas es algo cultural, no sólo de la cultura oficial proveniente del sistema educativo, sino además de la cultura antropológica y cultura popular.
Un ejemplo lo tenemos en EEUU, que además de contener algún que otro fanático creacionista, tiene una población que apoya la ciencia. Una manera de demostrar esto mismo es la cantidad de proyectos participativos que surgen allí para que la población en general aporte su granito de arena a la ciencia.
Hay proyectos como el que estudia las abejas de la zona en la que se vive y así saber sobre la ecología local y la evolución del colapso de las colmenas, pudiéndose elaborar mapas al respecto. Hay otro que ha solicitado a aficionados a la jardinería datos sobre fechas de floración y desarrollo de las plantas para ver cómo está evolucionando el cambio climático. Galaxy Zoo, otro proyecto participativo, permite a sus voluntarios clasificar galaxias procedentes de un catálogo astronómico.
El que sin duda abrió la veda en este tipo de proyectos fue Seti at Home que usa tiempo de CPU, en momentos de desuso de los ordenadores, para el análisis de datos provenientes de señales radioléctricas en busca de señales extraterrestres. Las personas interesadas sólo tenían que instalar un programa especial que funcionaba a modo de salvapantallas. Hubo otro proyecto, basado en el mismo concepto informático, para el estudio de fármacos y hay otro para encontrar números primos de Mersenne aún en curso.
Un subproducto salido de esa idea es EvoGrid. Si Seti at Home no parece proporcionar pruebas de vida inteligente, ¿por qué no buscar el origen de la vida aquí mismo, en una granja de ordenadores de sobremesa o con miles de computadores conectados en red? Ver cómo evoluciona una vida artificial en directo puede ser muy interesante e instructivo.
Este mes un equipo de investigadores de Silicon Valley planea transformar parte del software originalmente diseñado para la búsqueda de vida extraterrestre de Seti at Home en uno que simule vida artificial en clusters de ordenadores.
El proyecto es la tesis doctoral de Bruce Damer, un experto en computación de Silicon Valley que ha desarrollado software para la NASA en la compañía Digital Space (Santa Cruz, California), y Peter Newman, un ingeniero informático. Tienen como meta encontrar pruebas de comportamiento autoorganizativo en simulaciones informáticas masivas que corran en muchas CPU simultáneamente. De este modo se trata de explicar la aparición de la vida en el mundo físico o cómo lo hizo en la Tierra.
El sistema de reconocimiento de patrones usado para Seti at Home parece ser una herramienta muy buena en esta búsqueda.
La vida digital artificial está basada en una idea original de los matemáticos John von Neumann y Stanislaw Ulam, cuando ambos trabajaban en Los Alamos Laboratory en los años cuarenta. El húngaro contribuyó a la primera bomba atómica y el polaco principalmente a la bomba H, pero también pensaban en otros asuntos y proyectos de investigación. Al primero se le ocurrió la idea del autómata celular, una cuadrícula de elementos capaces de estar en distintos estados que se adquieren bajo reglas sencillas y que se puede programar fácilmente. El «juego de la vida» de Conway es un buen ejemplo de esto.
Más tarde, con el advenimiento del hardware potente se dieron pasos más sofisticados. Thomas Ray creo «Tierra», que despertó gran atención a principios de los noventa. El programa, que corría en más de 100 estaciones de trabajo, demostró que ciertos aspectos de la evolución biológica se podían simular digitalmente. Más recientemente se ha comercializado Spore, un juego que explora este tipo de ideas en plan lúdico.
A pesar del interés de este tipo de sistemas, el modelado de mundos «biológicos» de juguete es un campo que no escapa a la crítica, porque la creación de formas digitales de vida no tiene comparación con la inmensa complejidad de los procesos biológicos reales.
Según George Dyson, historiador científico entrevistado por el New York Times, cada 10 años se hace revivir este tipo de ideas. Aunque otros, como Richard Gordon de University of Manitoba, apoyan este tipo de esfuerzos.
Damer, respondiendo a los escépticos, dice que acoplando un mayor número de sistemas computacionales entre sí de tal modo que conste de miles de CPU, EvoGrid podría detectar la emergencia de la vida. El principal desafío según él no es la generación de algún tipo de interacción molecular novedosa, sino el análisis de lo que está sucediendo.
Para la construcción de EvoGrid los investigadores se apoyan en dos proyectos open-source. El primero es Boic, que es un sistema (financiado por la NSF) que usa Internet para que los científicos aprovechen los periodos de baja actividad computacional de ordenadores conectados al sistema. La semana antepasada, por ejemplo, el sistema estaba compuesto por 500.000 ordenadores que generaron 2,45 petaflops de capacidad de cómputo. Por el contrario en junio pasado la computadora más potente del mundo construida por IBM en Los Alamos «sólo» llegó a los 1,1 petaflops.
Además, para esta evolución digital de la vida, EvoGrid usa un segundo programa denominado Gromacs, desarrollado por la Universidad de Groningen (Holanda) que simula la interacción molecular.
La idea es que EvoGrid sea capaz de simular las condiciones químicas de los océanos primitivos y así crear una «sopa primordial digital» que evolucione a formas más complejas.
De momento, o al menos así lo parece, no planean dejar que participen voluntarios con sus ordenadores caseros, pero ¿quién sabe? Seguro que en algunos sitios hay gente dispuesta a hacerlo.
Fuentes y referencias:
Noticia en el New York Times.
Web de Evogrid.
2 Comentarios
RSS feed for comments on this post.
Lo sentimos, esta noticia está ya cerrada a comentarios.




lunes 5 octubre, 2009 @ 6:22 pm
Excelente nota, gracias por compartirla, estaré haciendo un post en mi blg tambien. Saludos.
miércoles 28 octubre, 2009 @ 12:27 pm
¿Puede un aficionado hacer un gran descubrimiento científico? Si pensamos en Gregor Mendel, un simple fraile jardinero, seguro que si. Voy a realizar esta mini-revisión temática:
«Grandes descubrimientos científicos hechos por aficionados».Creo que aparte del mencionado genio popular, habría otros. Es la hipótesis teórica que voy a probar.