Selección prebiótica
Proponen un escenario en el que la selección natural pudo operar a las escala bioquímica para reducir la entropía y aumentar la complejidad del sistema, lo que permitiría más tarde la aparición de la vida en la Tierra.
|
La génesis de la vida en la Tierra es una de los grandes misterios. Entre las moléculas prebióticas simples generadas en la Tierra primitiva y la primera célula hay una gigantesco océano de ignorancia. Esta es una de las razones por las que se propuso en su día la panspermia, que no soluciona el problema, sino que lo traslada.
Una vez se dispone de la primera célula es muy fácil explicar la vida terrestre, su complejidad y la historia que ha sufrido a lo largo de estos 3800 millones de años. La evolución darwiniana proporciona las herramientas para ello. Pero no es fácil aplicar estas reglas darwinistas a la evolución bioquímica prebiótica.
Ahora,un estudio realizado por físicos de la Universidad Ludwig-Maximilians de Munich demuestra que las características fundamentales de las moléculas monopoliméricas son suficientes para permitir procesos de selección en un esquema prebiótico plausible.
Antes de que surgiera la vida en la Tierra había numerosos procesos fisicoquímicos altamente caóticos en nuestro planeta. Había toda una plétora de pequeños compuestos y polímeros de longitud muy variada hechos a partir de subunidades, como puedan sea las bases que hay en el ADN y ARN. Estos compuestos se combinaban de multitud de maneras.
Antes de que emergieran procesos químicos análogos a los procesos biológicos, el nivel de desorden en este sistema tenía que reducirse. En este estudio liderado por Dieter Braun se muestran las aspectos básicos de los polímeros simples junto a ciertos aspectos del ambiente prebiótico que pudieron dar lugar a procesos de selección que redujeran el desorden.
En una publicación previa, este grupo de investigadores exploró cómo el orden espacial pudo haberse desarrollado en las pequeñas cámaras rellenas de agua de las rocas volcánicas porosas en el fondo del océano. En este estudio que nos ocupa ahora se muestra cómo las hebras de ARN pueden acumularse localmente en grandes cantidades gracias a la presencia de diferencias de temperatura y a un fenómeno convectivo conocido como efecto Soret.
«El problema es que las secuencias de bases de las moléculas más largas que se obtienen son totalmente caóticas», dice Braun.
Las ribozimas evolucionadas (enzimas basadas en ARN) tienen una secuencia de bases muy específica que permite que las moléculas se plieguen en formas particulares, mientras que la gran mayoría de los oligómeros formados en la Tierra Primitiva probablemente tenían secuencias aleatorias.
«El número total de posibles secuencias de bases, conocido como el espacio de secuencia, es increíblemente grande. Esto hace que sea prácticamente imposible ensamblar las estructuras complejas características de las ribozimas funcionales o moléculas comparables mediante un proceso puramente aleatorio», dice Patrick Kudella. Esto llevó a este equipo de investigadores a sospechar que la ampliación de moléculas para formar oligómeros más grandes estaba sujeta a algún tipo de mecanismo de preselección.
Anteriormente al momento del origen de la vida solo había unos pocos procesos físicos y químicos muy simples en comparación con los sofisticados mecanismos de replicación de las células, por lo que la selección de secuencias debe basarse en el entorno y en las propiedades de los oligómeros. Aquí es donde entra la investigación del grupo de Braun.
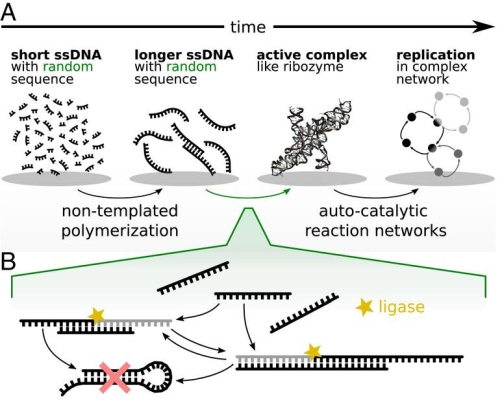
Para la función catalítica y la estabilidad de los oligómeros, es importante que se formen cadenas dobles como la conocida estructura helicoidal del ADN. Esta es una propiedad elemental de muchos polímeros y permite complejos con partes de cadena doble y simple.
Las partes monocatenarias se pueden reconstruir mediante dos procesos. El primero mediante la denominada polimerización, en la que las hebras se completan con bases simples para formar hebras dobles completas. El otro es mediante lo que se conoce como ligadura. En este proceso, se unen oligómeros más largos. Aquí, se forman partes de cadena doble y monocatenaria, lo que permite un mayor crecimiento del oligómero.
«Nuestro experimento comienza con una gran cantidad de cadenas de ADN cortas y en nuestro modelo para oligómeros tempranos usamos solo dos bases complementarias: adenina y timina. Suponemos que la ligadura de cadenas con secuencias aleatorias conduce a la formación de cadenas más largas, cuyas secuencias de bases son menos caóticas», dice Braun.
Luego, el grupo de Braun analizó las mezclas de secuencias producidas en estos experimentos utilizando un método que también se usa para analizar el genoma humano. La prueba confirmó que la entropía de la secuencia, es decir, el grado de desorden o aleatoriedad dentro de las secuencias recuperadas, se redujo de hecho en estos experimentos.
Los investigadores también pudieron identificar las causas de este orden autogenerado. Descubrieron que la mayoría de las secuencias obtenidas se dividían en dos clases, con composiciones de bases con un 70% de adenina y un 30% de timina, o viceversa.
«Con una proporción significativamente mayor de una de las dos bases, la hebra no se puede plegar sobre sí misma y permanece como un socio de reacción para la ligadura», explica Braun. Por tanto, apenas se forman en la reacción hebras con la mitad de cada una de las dos bases.
«También vemos cómo pequeñas distorsiones en la composición de la reserva de ADN corto dejan patrones con patrones distintos que dependen de la posición, especialmente en cadenas largas», dice Braun. El resultado sorprendió a los investigadores, porque una hebra de solo dos bases diferentes con una proporción de bases específica tiene formas limitadas de diferenciarse entre sí. «Sólo los algoritmos especiales pueden detectar detalles tan sorprendentes», dice Annalena Salditt, coautora del estudio.
Estos experimentos muestran que las características más simples y fundamentales de los oligómeros y su entorno pueden proporcionar las bases para procesos de selección. Incluso en un modelo simplificado pueden entrar en juego varios mecanismos de selección que tienen un impacto en el crecimiento de la hebra a diferentes escalas de longitud y son el resultado de diferentes combinaciones de factores.
Según Braun, estos mecanismos de selección eran un requisito previo para la formación de complejos catalíticamente activos como las ribozimas y, por lo tanto, desempeñaron un papel importante en el surgimiento de la vida desde el caos.
Copyleft: atribuir con enlace a https://neofronteras.com
Fuentes y referencias:
Artículo original.
Esquema: Proceedings of the National Academy of Sciences.
4 Comentarios
RSS feed for comments on this post.
Lo sentimos, esta noticia está ya cerrada a comentarios.




viernes 26 febrero, 2021 @ 7:53 pm
Un resultado prometedor al conseguir hebras con menor entropía que las iniciales.
De cara a plantear el origen de la vida, no hay que olvidar que las hebras no se autoensamblan solas: se requiere que un catalizador para que se produzca la reacción, en este caso una enzima (ADN-ligasa).
sábado 27 febrero, 2021 @ 10:54 am
No se olvidan de la ligasa en la ilustración. A mi lo que me parece más difícil es que estos fenómenos caóticos se produjesen en pequeñas celdas donde la temperatura puede decirse que no tiene gradientes, los cuales son necesarios para el efecto Soret del que tengo un concepto bastante impreciso. De todas formas, tenemos la fuente de calor (= energía) precisa para esa posible disminución de entropía. No me resulta fácil comprender el artículo.
domingo 28 febrero, 2021 @ 2:07 am
Si no entiendo mal, han comparado los resultados cuando aplican cierto ciclo de calentamiento y cuando no lo aplican:
«To test templated elongation of polymers in pools of random sequence oligomers, we prepared a 10 µM solution of 12-mer DNA strands composed with nucleobases A and T (sequence space: 4,096) and subjected it to temperature cycling, similar to ref. 21 with 20 s at denaturation temperature of 75 °C and 120 s at ligation temperature of 33 °C. Temperatures were selected according to the melting dynamics of the DNA pool; the time steps were prolonged relative to Toyabe and Braun (21) (SI Appendix, section 5.3) because of a greater sequence space. The larger sequence space of full random 12-mers with all four bases did not show any ligation under the same experimental conditions (SI Appendix, section 5.2). The sample was split into multiple tubes and exposed to 200, 400, 600, 800, and 1,000 temperature cycles, and one tube was kept at 4 °C for reference, all without influx or outflux of strands. Fig. 1D suggests the maximum depletion of the original 12-mer pool after 1,000 temperature cycles was only about 31%.
To study the length distributions in our samples, we used polyacrylamide gel electrophoresis (PAGE, Fig. 1D). The first lane is the reference sequence not exposed to temperature cycling, where small amounts of impurities are visible at short lengths (SI Appendix, section 3.1). The latter lanes show the temperature-cycled samples. As the number of cycles increases, progressively longer strands in multiples of 12 emerge, because the original pool only consisted of 12-mers. Fig. 1C shows the concentration quantification of each lane (compare SI Appendix, section 3). For higher cycle counts, the total amount of products increases, and the concentration as a function of length decreases slower. The behavior of this system is dependent on the time and temperature for both steps in the temperature cycle, the monomer-pool concentration, and the sequence space of the pool (SI Appendix, section 5).
domingo 28 febrero, 2021 @ 10:14 am
Bueno, a mí no me parece tan poco un 35 % tras 1000 ciclos, ya que la naturaleza tenía todo el tiempo de mundo. Y aunque luego habla de las hebras múltiplos de 12, también hace referencia a la temperatura y al tiempo, y esto último me parece importante.
Gracias por tu ayuda y un fuerte abrazo.