Memoria genética-bacteriana
Un nuevo sistema permite insertar la información del ambiente que se desee en el ADN de una colonia bacteriana que hace las veces de memoria distribuida.
|
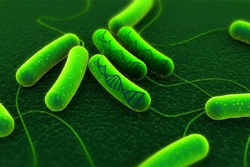
En un paso más de la biología sintética, un grupo de investigadores del MIT proponen un sistema mediante el cual células bacterianas modificadas pueden actuar de sistemas robóticos grabadores que recolectan información y la almacenan en su ADN.
Estas células podrían usarse como sistema de vigilancia de la calidad de las aguas, para medir la cantidad de azúcar que una persona ingiere, etc. Así por ejemplo, se podría tener unas bacterias viviendo en nuestro tracto digestivo durante días o meses y al cabo de ese tiempo recuperar unas cuantas y saber lo que ha ido pasando ahí dentro en ese tiempo.
Desde hace tiempo existen sistemas biológicos sintéticos capaces de efectuar las operaciones lógicas que efectúan los computadores, pero el almacenamiento de la información obtenida era un escollo para el desarrollo de este tipo de dispositivos. Hay ya varios sistemas de memoria en estos sistemas, pero estos métodos requieren un gran número de elementos regulatorios que limitan la cantidad de información que puede ser almacenada.
Según Timothy Lu, construir circuitos genéticos no sólo requiere de computación y lógica, sino además de almacenamiento de información y el ADN proporciona una forma de memoria muy estable que nos da la capacidad de efectuar tareas computacionales complejas.
En trabajos previos el almacenamiento de datos en este tipo de sistemas era muy laborioso, pero Lu y Fahim Farzadfard describen en su último artículo cómo grabar muchos tipos de datos simultáneamente y registrar entradas de datos en el tiempo. Además describen cómo leer linealmente en el tiempo esa información y secuenciarla en ADN. Al método lo denominan SCRIBE (Synthetic Cellular Recorders Integrating Biological Events).
El trabajo de investigación de este equipo empezó hace tres años en un intento de proyecto de edición en la que las células podrían ser modificadas para incorporar información a sus genomas.
El genoma bacteriano está compuesto por ADN, pero al tratarse de células procariotas no se encuentra en el núcleo, sino “disuelto” en el citoplasma celular. Es posible insertar nuevos genes en este genoma usando, por ejemplo, enzimas procedentes de virus (recombinasas) sobre las pocas hebras simples de ADN que hay (el resto es de doble hebra). El sistema no es muy efectivo porque las bacterias no poseen muchas hebras simples, pero hace ya bastante tiempo estos dos investigadores encontraron que una bacteria del suelo fabricaba grandes cantidades de hebras de ADN simples gracias una estructura flotante denominada retrón.
No se sabe la función natural de los retrones, pero Farzadfard y Lu se dieron cuenta de que podía reprogramarlos para producir hebras simples de ADN que codificara información que se quisiera usando una enzima viral y almacenándola en el genoma bacteriano.
En su último trabajo describen la creación de una colonia de E. coli en la que las bacterias contienen retrones que reaccionan a la presencia de un compuesto químico y provocan un cambio genético que las hace resistentes a un antibiótico. Además, cuanto mayor es la presencia del químico mayor es la proporción de células resistentes a los antibióticos que resultan, por lo que es una manera de medir la concentración.
La recombinasa puede insertar una secuencia específica en el lugar del genoma que se desee y su información se almacena durante todo el periodo de vida de la colonia, pasando de generación en generación. Este ADN es insertado en partes no funcionales del genoma para que no afecte a la bioquímica normal de la bacteria, pero su información puede ser leída mediante la secuenciación del genoma.
En el caso de la resistencia al antibiótico, se usó esta característica porque puede ser empleada fácilmente como marcador en ese modelo de prueba de concepto. Añadiendo el antibiótico se puede ver qué células en cuestión (las que sobreviven) habían adquirido esa capacidad y, por tanto, poseían la información registrada que se pretendía. De este modo, se ahorraban secuenciaciones de genoma.
SCRIBE complementa otros métodos existentes, pero, a diferencia de otros intentos anteriores en los que los métodos eran similares a una memoria digital, la memoria de este nuevo método es más bien analógica y no está contenida en una célula, sino en toda la colonia, se trata de una memoria distribuida a lo largo de toda la población en lo que constituye un poderoso método de computación, según los investigadores. Cada célula no es un “disco duro”, sino que lo es la población.
Esta memoria colectiva puede revertirse y reescribirse de nuevo mediante luz gracias a la inserción en la “circuitería” genética de proteínas fotosensibles. Además, estos investigadores fueron capaces de grabar dos variables a la misma vez usando la inserción de secuencias distintas de ADN y creen que la técnica, aunque de momento ineficiente, se puede escalar para realizar tareas más complejas.
Usando un método controlado por la iluminación los investigadores pudieron desarrollar un sistema que detectase la luz a base de un metabolito de la lactosa denominado IPTG y un antibiótico, pero se podría hacer algo similar con muchas otras moléculas y señales, incluidas señales químicas producidas por células.
Este método se podría usar en sensores de vigilancia ecológica, como la medida de acidez, concentración de dióxido de carbono o contaminantes. Se podría emplear en aplicaciones médicas. Además de la ya mencionada al principio, se podrían usar células de este tipo como marcadores.
Pero también se podría usar en sistemas computacionales biológicos para resolver problemas en los que se requiriera un alto nivel de proceso en paralelo, pues bastaría usar miles de millones de bacterias de fuerza bruta para resolverlos y todo a un coste energético mínimo.
Copyleft: atribuir con enlace a http://neofronteras.com/?p=4544
Fuentes y referencias:
Artículo original
Ilustración: Christine Daniloff/MIT.
1 Comentario
RSS feed for comments on this post.
Lo sentimos, esta noticia está ya cerrada a comentarios.




lunes 17 noviembre, 2014 @ 11:02 am
Sólo una confirmación: Cuando dice «hebras simples» se refiere a una sola cadena de fosfato-desoxiribosa con su «medios peldaños» de adenina, citosina, etc. La «doble hebra» es la «cadena-escalera helicoidal» tal como suele representarse el ADN. Casi estoy seguro de que es así, pero agradecería una certificación. Es que mi cultura celular es muy superficial.