Nuevo algoritmo para genes, lenguas o terroristas
Gracias a ciertos algoritmos se pueden encontrar subgrafos dentro de redes genéticas que ayuden al entendimiento de ciertas enfermedades. También se pueden aplicar a las redes sociales en Internet.
|
Las enfermedades humanas, las estructuras lingüísticas, el terrorismo y las redes sociales parecen tener poco que ver. Sin embargo, éstas tienen en común que descansan sobre una red. Analizando el grafo de una red de este tipo se pueden encontrar las subestructuras sutiles que contiene como, por ejemplo, las comunidades contenidas en una red social.
En un artículo reciente (Physical Review E 77:016104 (2008)) Weixiong Zhang y Jianhua Ruan, de Washington University publican un algoritmo que automáticamente identifica comunidades y estructuras sutiles en varios tipos de redes.
Muchos sistemas complejos pueden ser entendidos como si fueran redes, como las redes sociales que hay en Internet o incluso las redes genéticas que estos investigadores estudian.
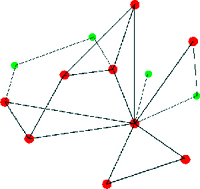
Desde el punto matemático estas redes pueden ser representadas como grafos. De este modo los objetos, como por ejemplo individuos de una red social, serían los puntos del grafo denominados vértices. Éstos estarían conectados por líneas llamadas aristas, si los individuos que unen están relacionados (ver dibujo). Si un grupo de individuos de una red social están fuertemente relacionados entre ellos y débilmente con el resto formarán una subred social que en teoría podría ser identificada como un subgrafo dentro del grafo general. Pero esta identificación requiere de un algoritmo eficiente que lo consiga dentro del marasmo general.
Las comunidades son relativamente independientes unas de otras, pero los investigadores piensan que cada comunidad podría corresponder a una unidad funcional fundamental. De este modo una comunidad en una red genética contiene usualmente genes con funciones similares, y de la misma manera una comunidad de la WWW frecuentemente enlazará a páginas web de temas similares.
Todo lo que Zhang y Ruan necesitan son datos. Su algoritmo es más escalable que otros similares que ya existen y pueden detectar comunidades a una escala más pequeña con alta precisión. Una de las ventajas de contar con esta herramienta matemática es su aplicación en biología computacional, sobre todo en el campo de la genética. Gracias a esto los investigadores podrían encontrar más fácilmente y entender mejor conjuntos (comunidades) de genes y sus redes asociadas, así como su cooperación a la hora de desencadenar una enfermedad, desde facilitar una infección vírica o hasta producir Alzheimer. Los investigadores ya lo han aplicado a la identificación de las comunidades genéticas de una red genes relacionados con la sepsis bacteriana.
Según estos investigadores, en redes sociales este algoritmo puede determinar cómo interacciona la gente en este tipo de estructuras como, por ejemplo, cómo los científicos colaboran unos con otros en su tema de investigación.
Otro campo de aplicación podría ser la detección de células terroristas. Accediendo a los datos de correo electrónico y similares se pueden detectar candidatos a esta clase de grupos e incluso saber su estructura jerárquica.
En sistemas biológicos hay montones de comunidades en las que muchas proteínas que están relacionadas entre sí para formar complejos. Se pueden usar este tipo de herramientas para identificar estructuras que estén embebidas en los datos disponibles. Así por ejemplo, estos investigadores han conseguido identificar la subestructura de tres complejos de ARN polimerasa distintos, que son cruciales para la transcripción genética, a partir de datos con mucho ruido.
La meta de estos investigadores es usar técnicas computacionales para resolver problemas biológicos y aquellos relacionados con enfermedades humanas. Uno de sus éxitos fue mostrar que casi todos los genes microARN intergénicos en humanos, ratones, arroz y Arabidopsis son transcritos por la ARN polimerasa II, que transcribe genes codificadores de proteínas. El microARN son pequeños trozos de ARN no codificante que regulan el desarrollo y la respuesta al estrés en casi todos las especies eucariotas que se han estudiado.
También consiguieron desarrollar un algoritmo llamado WordSpy que identificó los elementos regulatorios-cis a partir de una gran número de secuencias genéticas.
WordSpy se inspira en una vieja técnica denominada estenografía, que se remonta a la Grecia clásica. El método no sólo permite analizar las secuencias genéticas, sino además las lenguas humanas. Este método ha sido aplicado, por ejemplo, para segmentar palabras y frases en el idioma chino.
Estos investigadores esperan usar estas herramientas algorítmicas para así comprender mejor la respuesta al estrés vírico en plantas como el arroz, o comprender mejor el desarrollo de la enfermedad de Alzheimer, el herpes, la hipertrofia cardíaca, el cáncer de pulmón, etc.
Y es que la matemática está en muchos sitios, aunque no lo queramos ver.
Fuentes y referencias:
Nota de prensa en Washington University
Propagación de cotilleos en redes sociales.
Splitting Terrorist Cells.
Math latest weapon in war on terror.
AMS.
3 Comentarios
RSS feed for comments on this post.
Lo sentimos, esta noticia está ya cerrada a comentarios.




jueves 27 marzo, 2008 @ 3:31 pm
«Y, es que la matemática está en muchos sitios,aunque no la queramos ver». Me imagino que más que no quererla ver, es que no podemos verla, por desconocimiento; evidentemente. No hace mucho leí una muy interesante con Roger Penrose en la que decía que las matemáticas tienen existencia por sí mismas, pero no en el sentido de la «existencia matemática» -que no siempre se traduce por «realidad física», sino que existen, es decir tienen una vida propia al margen de nuestra propia existencia. El entrevistador quedó un tanto desconcertado, y el propio Penrose, supongo que para confortarle, le dijo «sí ya sé que suena un poco desconcertante, pero…». Realmente, creo que todo lo que contiene el Universo, son matemáticas (aunque impresionen o den miedo al común de los mortales).
miércoles 2 abril, 2008 @ 7:54 pm
Parece entonces que Penrose se quedó en Platón desde un punto de vista filosófico. Aunque para poder contestar a este tipo de dudas quizás el único punto de vista que tengamos sea precisamente el filosófico.
sábado 10 mayo, 2008 @ 1:21 pm
Muy interesante. Para quienes estén interesados en la biocomputación dejo un enlace a artículos traducidos al español (traducciones de calidad desigual) de la revista PLoS Computational Biology.